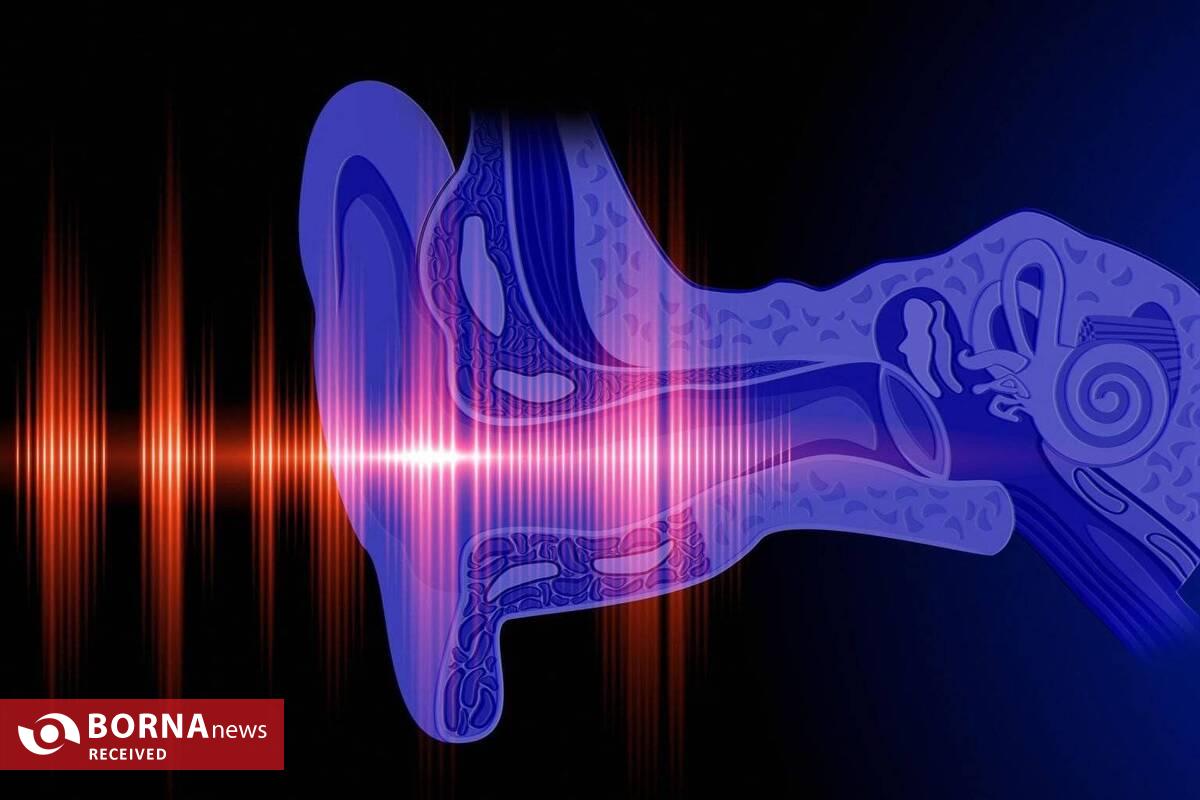
کشف پیوند ژنتیکی اوتیسم با بیماری نادر عضلانی

پژوهشگران بیمارستان کودکان سیککیدز در کانادا و دانشگاه نوادا در لاسوگاس موفق به شناسایی یک ارتباط ژنتیکی میان اختلال طیف اوتیسم (ASD) و بیماری نادر ارثی «دیستروفی میوتونیک نوع یک» (DM۱) شدند.
این یافته که در نشریه Nature Neuroscience منتشر شده، نظریه دیرینه نقش اصلی «از دست رفتن عملکرد ژن» را در اوتیسم به چالش میکشد و سازوکاری متفاوت را برای بروز رفتارهای اجتماعی مرتبط با اوتیسم پیشنهاد میکند.
DM۱ نوعی بیماری ژنتیکی است که با ضعف پیشرونده و تحلیل رفتن عضلات شناخته میشود. با اینکه اوتیسم حدود یک درصد از جمعیت عمومی را درگیر میکند، احتمال تشخیص آن در افراد مبتلا به DM۱، ۱۴ برابر بیشتر است.
تکرارهای DNA و اختلال در فرآیند "اسپلایسینگ"
محققان دریافتند که جهش ژنتیکی مسئول DM۱، یعنی تکرارهای دنبالهدار (Tandem Repeat Expansions یا TREs) در ژن DMPK، باعث اختلال در رشد طبیعی مغز نیز میشود. این تکرارها عملکرد فرآیندی حیاتی به نام "اسپلایسینگ ژن" را بر هم میزنند؛ فرایندی که برای بیان صحیح ژنها ضروری است. در نتیجه، تعادل تولید پروتئین در مغز به هم میریزد و فرآیند اسپلایسینگ تعداد زیادی از ژنهای مؤثر در عملکرد مغز دچار اختلال میشود. به باور دانشمندان، این تغییرات مولکولی ممکن است با بروز ویژگیهای اجتماعی و رفتاری مرتبط با اوتیسم در افراد مبتلا به DM۱ ارتباط داشته باشد.
پروتئینهایی که در دام RNA سمی گرفتار میشوند
رایان یوان، زیستشناس ارشد در برنامه ژنتیک و زیستشناسی ژنومی در SickKids، در این باره میگوید: «یافتههای ما راه جدیدی برای توصیف توسعه ژنتیکی اوتیسم ارائه میدهند. با شناسایی مسیر مولکولی دخیل در این ارتباط، میتوانیم به سمت روشهای نوین تشخیص ASD و توسعه درمانهای هدفمند و دقیق حرکت کنیم.»
به گزارش ساینس دیلی، تکرارهای دنبالهدار زمانی ایجاد میشوند که بخشهایی از DNA چندین بار تکرار شوند؛ با هر بار تکرار، احتمال بروز خطا در عملکرد ژن افزایش مییابد. یوان در سال ۲۰۲۰ بیش از ۲۵۸۸ محل متفاوت در ژنوم افراد مبتلا به اوتیسم را شناسایی کرد که در آنها TREs فراوانتر از حد معمول دیده میشدند.
لوکاش اشنایدر، استادیار و محقق ارشد در UNLV، در این باره میگوید: «یکی از این تغییرات در بیماری نادر عصبی-عضلانی برای من برجسته شد و همین آغازگر ارتباطدادن نقاط مختلف به هم بود. ما به پیوند مولکولیای رسیدیم که احتمالاً منشأ بروز علائم اوتیسم در کودکان مبتلا به دیستروفی میوتونیک است.»
مکانیسم اختلال: جذب پروتئینها توسط RNA سمی
با افزایش تکرارهای دنبالهدار در ژن DMPK، RNA تغییریافتهای تولید میشود که به پروتئینی حیاتی برای تنظیم فرآیند اسپلایسینگ در مغز متصل میشود. این RNA سمی، پروتئین را در خود جذب کرده و مانع از عملکرد طبیعی آن در دیگر بخشهای ژنوم میشود؛ موضوعی که به عدم تعادل پروتئینی و اختلال در اسپلایسینگ ژنهای دیگر میانجامد.
یوان میگوید: «TREها مانند اسفنجی عمل میکنند که پروتئینهای مهم را از ژنوم جذب میکند. بدون این پروتئینها، عملکرد سایر قسمتهای ژنوم مختل میشود.»
آزمایشگاههای یوان و اشنایدر اکنون در حال بررسی این هستند که آیا اختلال در اسپلایسینگ در ژنهای دیگری که با اوتیسم مرتبط هستند نیز رخ میدهد یا خیر. همچنین آنها در تلاشاند بفهمند که چگونه این یافتهها میتوانند به توسعه درمانهای دقیقتری برای آزادسازی این پروتئینها و بازگرداندن عملکرد طبیعی ژنها منجر شود.
بخشی از این تلاشها پیشتر آغاز شده است؛ بهطور مثال در سال ۲۰۲۰، کریستوفر پیرسون، زیستشناس ارشد در SickKids، مولکولی را شناسایی کرد که میتواند TREs را در بیماری هانتینگتون کوتاه کند. اگرچه کاربرد آن در سایر بیماریها هنوز نیاز به تحقیقات بیشتری دارد، تیم پژوهشی امیدوار است که این یافتهها بتواند مسیر پژوهشهای آینده و درمان شرایطی، چون DM۱ و ASD را متحول کند.
انتهای پیام/